G-GENOCOD : L’Intelligence Artificielle au service de la découverte de molécules
Publié par Nicolas Gutowski, le 21 mai 2025 1.1k
Imaginez un immense océan composé de milliards de molécules différentes. Parmi ces innombrables possibilités, seules quelques-unes sont véritablement exceptionnelles : capables de guérir des maladies ou encore de créer des matériaux révolutionnaires. Repérer ces molécules rares et prometteuses au milieu d'une immensité de possibilités représente un véritable casse-tête scientifique pour les chimistes.
Même en ne se limitant qu’aux molécules relativement simples, avec seulement jusqu’à 17 atomes lourds (carbone, oxygène, azote, soufre, halogènes), le nombre total de molécules envisageables atteint 1022, soit l'équivalent de 10 000 milliards de milliards de molécules (c.-à-d. 10 000 000 000 000 000 000 000). Tester expérimentalement ou simuler chacune de ces molécules est impossible en pratique, car cela prendrait des siècles de calculs sur des ordinateurs très puissants. De plus, certaines molécules, bien que théoriquement possibles, ne sont pas réalisables en laboratoire. Il est donc essentiel de disposer de stratégies capables d’explorer intelligemment cet immense « paysage moléculaire » pour identifier efficacement les composés les plus prometteurs tout en évitant de gaspiller des ressources précieuses sur des pistes peu pertinentes.
C’est précisément l’un de ces challenges que relève le projet G-GENOCOD (Graph-GEneration for NOvel Compound Discovery) : mettre à profit l’Intelligence Artificielle (IA) pour créer un laboratoire virtuel capable de générer, d’évaluer et de sélectionner automatiquement les molécules les plus intéressantes à fabriquer et à tester en laboratoire. Soutenu par le dispositif régional « Étoiles Montantes en Pays de la Loire », ce projet de 2 ans (2024 - 2025) est mené au laboratoire d’informatique LERIA de l’université d’Angers, en collaboration avec le laboratoire MOLTECH-Anjou qui est une Unité Mixte de Recherche (UMR 6200) associant le CNRS et l’Université d’Angers, spécialisée dans la conception et l’étude de matériaux moléculaires.
Un paysage moléculaire à explorer
Mais comment visualiser tout cela concrètement ? Pour simplifier la compréhension, on peut imaginer l’espace des molécules comme un relief montagneux quasi infini :
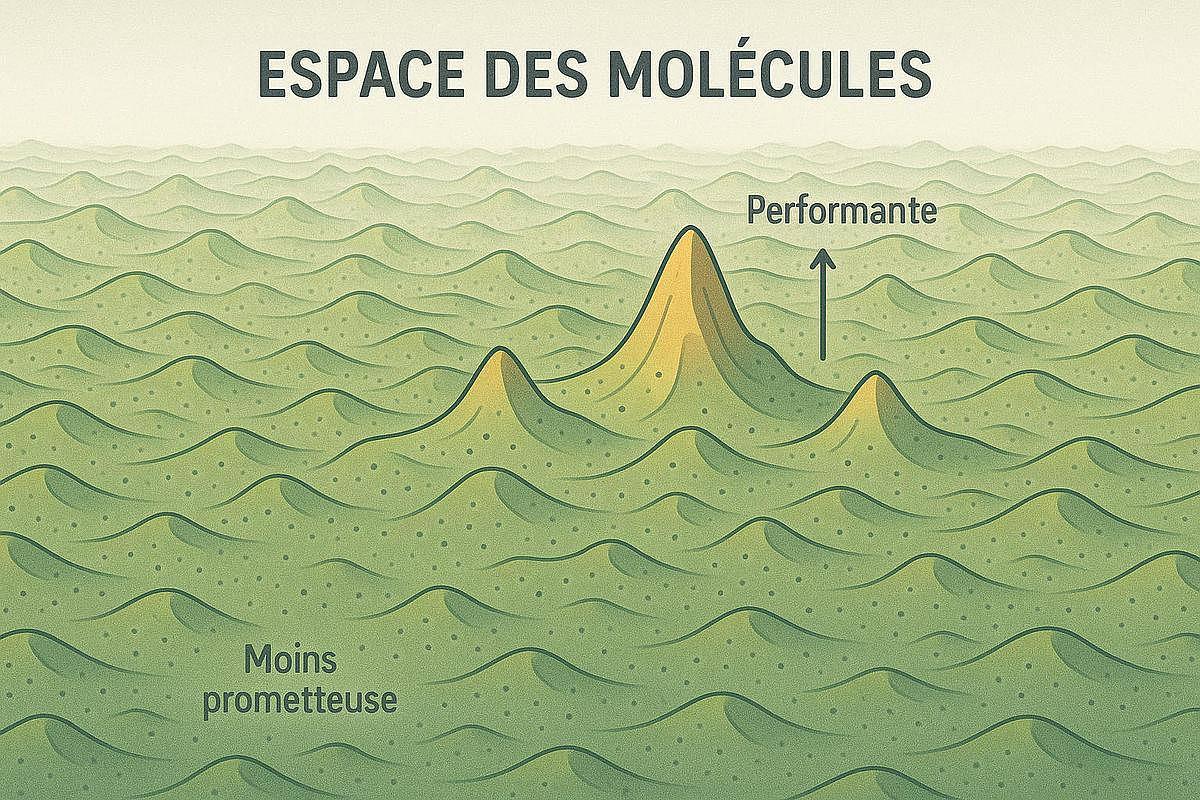
- Chaque point du terrain symbolise une molécule différente.
- Plus ce point est élevé, plus la molécule est performante selon un objectif donné, par exemple son efficacité thérapeutique ou sa facilité de synthèse.
- Les sommets désignent donc les molécules les plus intéressantes, tandis que les creux ou vallées correspondent à celles qui sont moins prometteuses.
Ainsi, explorer ce paysage revient à choisir les itinéraires les plus intelligents pour atteindre les meilleurs sommets sans perdre trop de temps à descendre dans des vallées infructueuses.
Un algorithme évolutionnaire : EvoMol
Pour explorer efficacement ce paysage complexe, le projet G-GENOCOD s’appuie notamment sur EvoMol , un algorithme qui imite les mécanismes de l’évolution naturelle pour générer de nouvelles molécules. Pour ce faire, il part d’un petit groupe de molécules simples (dites “précurseurs”), puis pour créer de nouvelles molécules candidates, il sélectionne à chaque étape les plus prometteuses et leur applique des modifications optimales, renforcées par les succès observés précédemment (ajout ou suppression d’atomes, modification des liaisons).
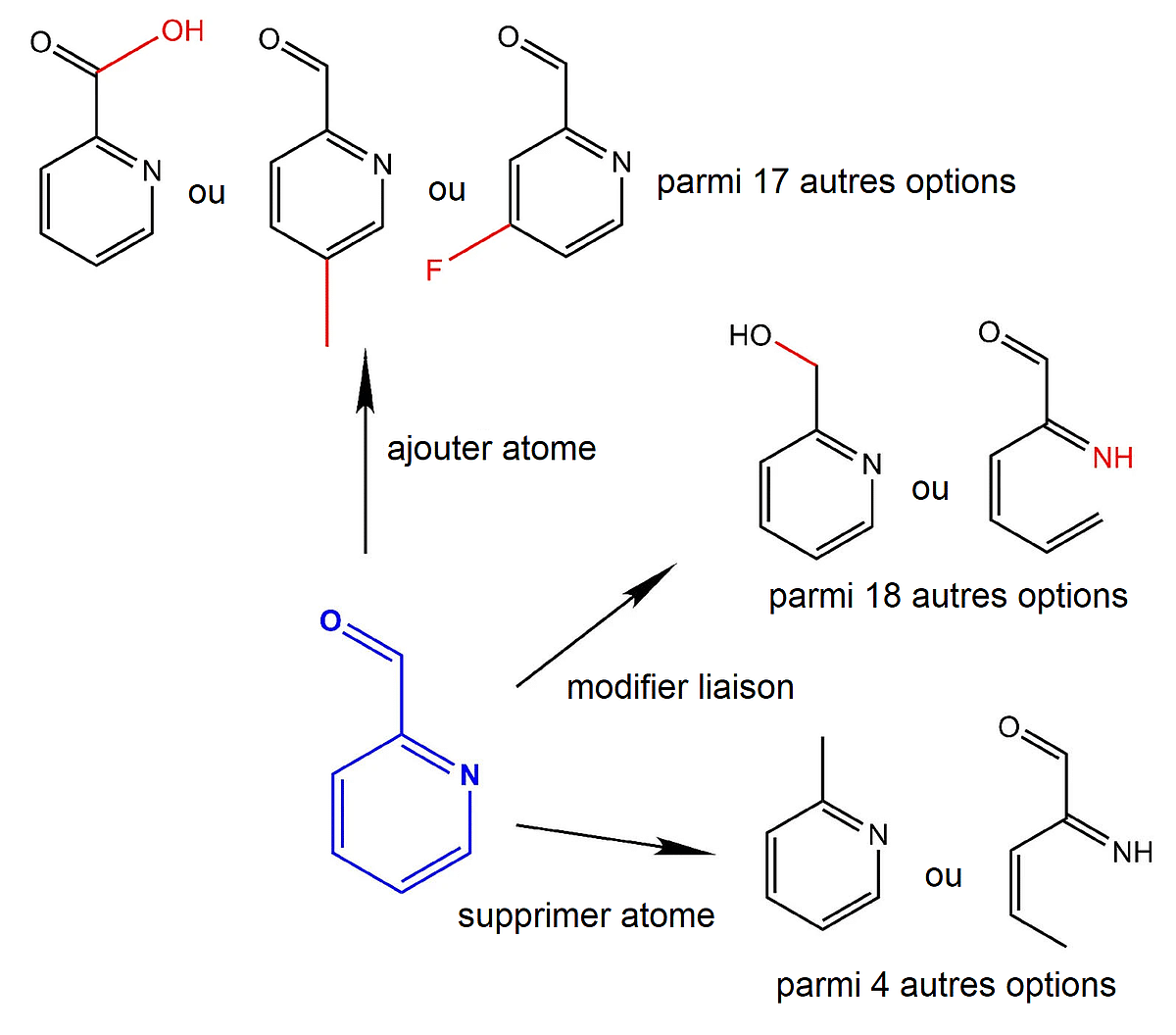
Grâce à cette méthode, il devient possible d’explorer efficacement l’espace moléculaire, en générant des molécules réalistes, fonctionnelles et chimiquement valides. Chaque étape du processus reste lisible, ce qui permet aux chercheurs de suivre et d’interpréter le cheminement des transformations tout au long du processus.
De la molécule à sa synthèse
Cependant, identifier une molécule prometteuse ne suffit pas : il faut également savoir comment la fabriquer en laboratoire. C’est l’un des seconds objectifs de G-GENOCOD qui intègre ProPreT5, un modèle basé sur l'architecture Transformer (modèle T5) entraîné pour prédire les produits de réactions chimiques. Ce modèle est entraîné à partir d’un grand nombre de réactions chimiques et peut prédire les produits chimiques obtenus à partir de réactifs donnés.
En s’appuyant sur des motifs réactionnels génériques — appelés templates — ce modèle permet de proposer, pour une molécule cible, des étapes de transformation plausibles à partir de précurseurs connus. L’objectif final du projet est d’utiliser les capacités de ProPreT5 au sein d’une recherche guidée par l’IA appelée MCTS (Monte-Carlo Tree Search), comparable à une exploration par essais successifs dans un arbre de décisions.
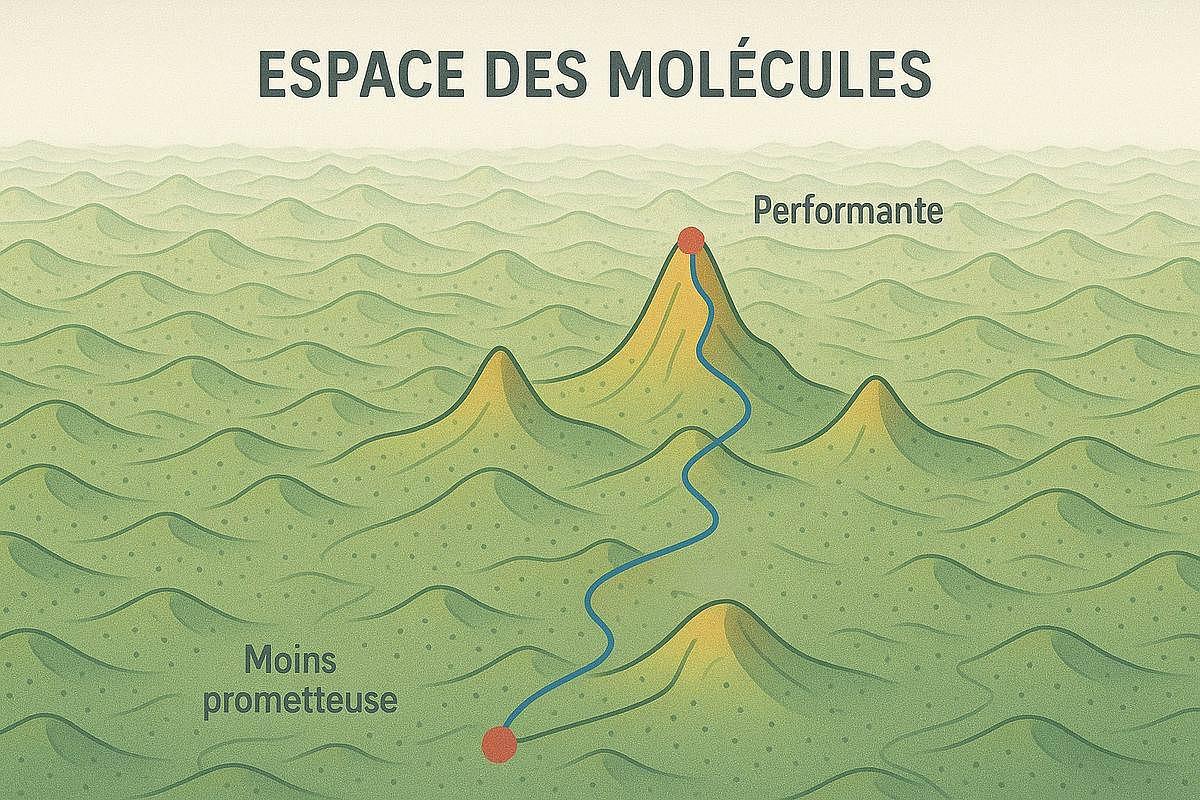
Pour bien comprendre son fonctionnement : Imaginez qu’en connaissance du sommet de notre montagne (la molécule cible), l’algorithme cherche à remonter à ce sommet à partir des vallées (les réactifs disponibles), en testant différents chemins possibles, étape par étape. Cette démarche permettrait d’indiquer aux chimistes des voies de synthèse plausibles et exploitables, rendant les propositions d’EvoMol directement actionnables au laboratoire MOLTECH-Anjou.
Des données représentatives pour une IA efficace
Cela dit, pour que ces méthodes soient vraiment efficaces, encore faut-il s’assurer de la qualité et de la représentativité des données utilisées. Or, certaines bases de données couramment employées pour entraîner les modèles d’IA sont trop spécialisées, ce qui limite l’application pratique dans le monde réel du fait de ces angles morts. Par exemple, des modèles entraînés uniquement sur des brevets industriels perdent en efficacité face à des réactions chimiques plus simples ou plus classiques, mais absentes de ces brevets.
C’est pourquoi G-GENOCOD accorde une attention particulière à développer des jeux de données plus diversifiés et plus représentatifs de la chimie réelle, ce afin de garantir que les modèles d’IA répondent plus efficacement aux besoins des chercheurs en chimie.
Conclusion
Ainsi, grâce au financement de la région Pays de la Loire et aux énergies scientifiques du territoire qui combinent toutes les approches innovantes décrites dans cet article, G-GENOCOD se constitue comme une plateforme complète pour accélérer radicalement la découverte de molécules nouvelles, efficaces et concrètement exploitables, ouvrant ainsi la voie à des avancées majeures dans le domaine de la santé, de l'énergie et des matériaux.